一般化線形混合モデル(GLMM)とは
一般化線形混合モデル (Generalized Linear Mixed Model; GLMM)とは、GLM をさらに発展させた統計モデルであり、観測できない個体差や場所差によるばらつきを表現することができます。
観測できない個体差や場所差というのは、例えば次のようなものが挙げられます。
- 痛覚の個人差
同じ痛みであっても人により反応は異なります。 - 採点の個人差
甘い採点者もいれば辛い採点者もいます。 - 地域差
地域によって性質のばらつきがあります。
これらの個体差や場所差を考慮していないモデルでは、過分散を引き起こしてしまいます。過分散とは、仮定した確率分布が想定している分散よりも大きな分散となっていることを意味します。ポアソン分布を例に過分散を表すと次の図のようになります。
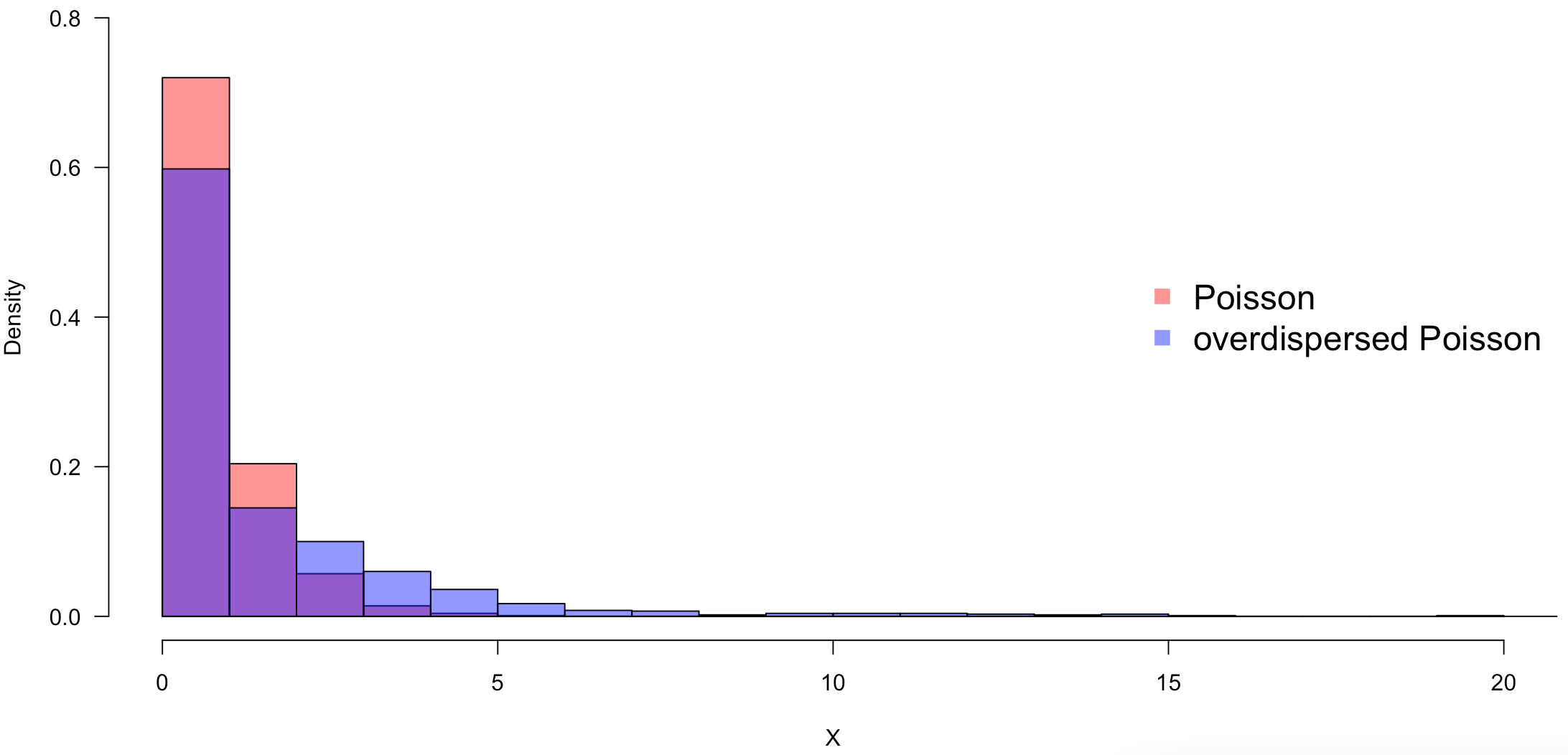
個体差や場所差が発生しているとoverdispersed Poissonのように本来のポアソン分布よりも大きなばらつきとなり、個体差や場所差を考慮しないままでは観測データを上手くモデリングすることができません。
GLMMでは、説明変数の全てを観測することは不可能と判断したうえで、個体差や場所差といった原因不明のばらつきをランダム効果として扱いモデリングします。
ランダム効果
GLMMでは線形予測子にランダム効果が追加されます。例えば、個体
ここで、ランダム効果
パラメータの最尤推定は
GLMM が必要なケース
GLMMが必要かどうかの判断基準は、同じ個体や場所から何度もサンプリングしているかどうかになります。例えば、個人に対して時間経過とともに繰り返し観測したデータであるパネルデータではGLMMがよく適用されます。
GLMM の構築
grouseticks というデータセットを使ってGLMMを構築します。grouseticksはアカライチョウのヒナの頭部に付着しているダニ数の観測データであり、次のカラムで構成されています。
INDEX: (factor) chick number (observation level)TICKS: number of ticks sampledBROOD: (factor) brood numberHEIGHT: height above sea level (meters)YEAR: year (-1900)LOCATION: (factor) geographic location codecHEIGHT: centered height, derived from HEIGHT
データの確認を行います。
> require(lme4)
> library(lme4)
> data(grouseticks)
> summary(grouseticks)
INDEX TICKS BROOD HEIGHT YEAR LOCATION cHEIGHT
1 : 1 Min. : 0.00 606 : 10 Min. :403.0 95:117 14 : 24 Min. :-59.241
2 : 1 1st Qu.: 0.00 602 : 9 1st Qu.:430.0 96:155 4 : 20 1st Qu.:-32.241
3 : 1 Median : 2.00 537 : 7 Median :457.0 97:131 19 : 20 Median : -5.241
4 : 1 Mean : 6.37 601 : 7 Mean :462.2 28 : 19 Mean : 0.000
5 : 1 3rd Qu.: 6.00 643 : 7 3rd Qu.:494.0 50 : 17 3rd Qu.: 31.759
6 : 1 Max. :85.00 711 : 7 Max. :533.0 36 : 16 Max. : 70.759
(Other):397 (Other):356 (Other):287
> head(grouseticks)
INDEX TICKS BROOD HEIGHT YEAR LOCATION cHEIGHT
1 1 0 501 465 95 32 2.759305
2 2 0 501 465 95 32 2.759305
3 3 0 502 472 95 36 9.759305
4 4 0 503 475 95 37 12.759305
5 5 0 503 475 95 37 12.759305
6 6 3 503 475 95 37 12.759305
> plot(TICKS ~ HEIGHT, las=1)
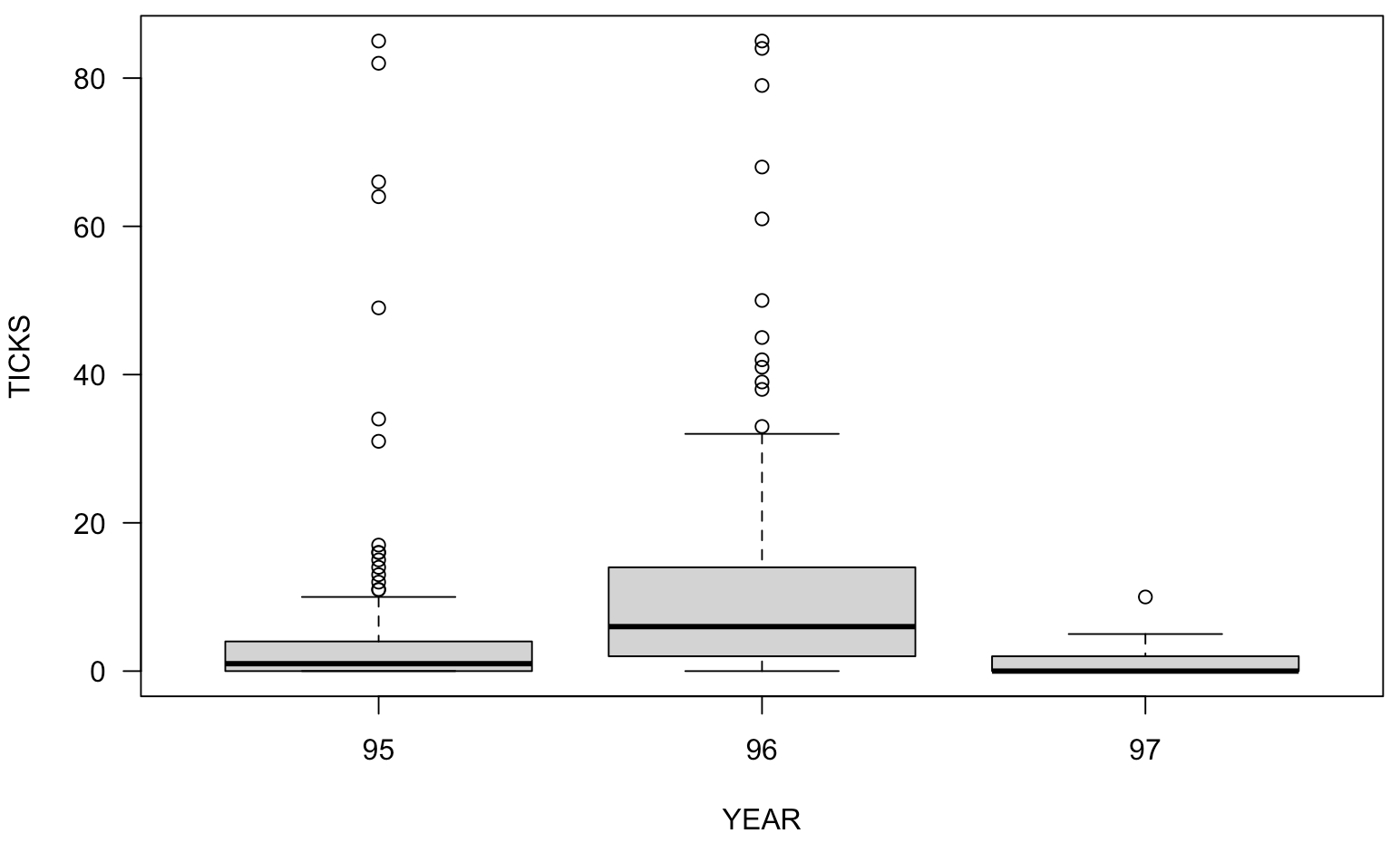
ポアソン回帰(ランダム効果なし)
今回はポアソン回帰モデルを構築します。平均のダニ数
ここで、Yearのダミー変数です。
次のコードでパラメータを推定することができます。
> fit_glm <- glm(TICKS ~ YEAR, family=poisson(), data=grouseticks)
> summary)(fit_glm)
Call:
glm(formula = TICKS ~ YEAR, family = poisson(), data = grouseticks)
Deviance Residuals:
Min 1Q Median 3Q Max
-4.7110 -2.8888 -1.5183 0.7139 17.1467
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 1.78318 0.03790 47.05 <2e-16 ***
YEAR96 0.62348 0.04492 13.88 <2e-16 ***
YEAR97 -1.64109 0.08977 -18.28 <2e-16 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
(Dispersion parameter for poisson family taken to be 1)
Null deviance: 5847.5 on 402 degrees of freedom
Residual deviance: 4549.4 on 400 degrees of freedom
AIC: 5486.4
Number of Fisher Scoring iterations: 6
AICは5486.4となっています。
ポアソン回帰(ランダム効果あり)
ランダム効果を考慮したポアソン回帰モデルを構築します。
Random intercept model
LOCATIONによる場所
このモデルは、ランダム効果が切片に入っていることからrandom intercept modelとも呼ばれます。
次のコードで TICKS ~ YEAR + (1 | LOCATION)のように記述します。
> fit_glmm <- glmer(TICKS ~ YEAR + (1 | LOCATION), family="poisson", data=grouseticks)
> summary(fit_glmm)
Generalized linear mixed model fit by maximum likelihood (Laplace Approximation) ['glmerMod']
Family: poisson ( log )
Formula: TICKS ~ YEAR + (1 | LOCATION)
Data: grouseticks
AIC BIC logLik deviance df.resid
2306.3 2322.3 -1149.1 2298.3 399
Scaled residuals:
Min 1Q Median 3Q Max
-6.6399 -0.8844 -0.4708 0.8543 5.5757
Random effects:
Groups Name Variance Std.Dev.
LOCATION (Intercept) 1.644 1.282
Number of obs: 403, groups: LOCATION, 63
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.64992 0.18319 3.548 0.000388 ***
YEAR96 0.94371 0.08492 11.113 < 2e-16 ***
YEAR97 -1.43642 0.12298 -11.681 < 2e-16 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Correlation of Fixed Effects:
(Intr) YEAR96
YEAR96 -0.295
YEAR97 -0.241 0.520
AICが2306.3となり、ランダム効果がないモデルよりも大きく改善されました。
LOCATIONのランダム効果の分散は1.644となっています。randf()関数で全てのLOCATIONのランダム効果を確認することができます。
> ranef(fit_glmm)
$LOCATION
(Intercept)
1 1.24539318
2 0.92293852
3 -0.93190888
.
.
.
61 -1.35144088
62 -0.95446024
63 -1.15277383
with conditional variances for “LOCATION”
1から63はLOCATIONの値です。1のinterceptは1.24539318、3のinterceptは-0.93190888となっています。解釈としては、LOCATION 1はLOCATION 3よりも相対的にダニの数が多いということになります。
さらにINDEXによるランダム効果
> fit_glmm <- glmer(TICKS ~ YEAR + (1 | LOCATION) + (1 | INDEX), family="poisson", data=grouseticks)
> summary(fit_glmm)
Generalized linear mixed model fit by maximum likelihood (Laplace Approximation) ['glmerMod']
Family: poisson ( log )
Formula: TICKS ~ YEAR + (1 | LOCATION) + (1 | INDEX)
Data: grouseticks
AIC BIC logLik deviance df.resid
1863.9 1883.9 -927.0 1853.9 398
Scaled residuals:
Min 1Q Median 3Q Max
-1.5003 -0.5933 -0.1154 0.2723 2.3743
Random effects:
Groups Name Variance Std.Dev.
INDEX (Intercept) 0.5081 0.7128
LOCATION (Intercept) 1.4679 1.2116
Number of obs: 403, groups: INDEX, 403; LOCATION, 63
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.2774 0.2107 1.316 0.188
YEAR96 1.2578 0.1821 6.906 4.97e-12 ***
YEAR97 -1.0856 0.2010 -5.400 6.67e-08 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Correlation of Fixed Effects:
(Intr) YEAR96
YEAR96 -0.536
YEAR97 -0.439 0.552
AICが1863.9となり、さらに改善されました。
LOCATIONのランダム効果の分散は1.4679、 INDEXのランダム効果の分散は0.5081となっています。INDEXの方がLOCATIONよりもランダム効果が小さいことが分かります。
Random coefficient model
LOCATIONによるランダム効果
このモデルは、ランダム効果が係数に入っていることからrandom coefficient modelとも呼ばれます。
次のコードで TICKS ~ YEAR + (YEAR | LOCATION)のように記述します。
> fit_glmm <- glmer(TICKS ~ YEAR + (YEAR | LOCATION), family="poisson", data=grouseticks)
> summary(fit_glmm)
Generalized linear mixed model fit by maximum likelihood (Laplace Approximation) ['glmerMod']
Family: poisson ( log )
Formula: TICKS ~ YEAR + (YEAR | LOCATION)
Data: grouseticks
AIC BIC logLik deviance df.resid
2266.7 2302.7 -1124.3 2248.7 394
Scaled residuals:
Min 1Q Median 3Q Max
-6.6442 -0.8112 -0.4797 0.8437 5.5779
Random effects:
Groups Name Variance Std.Dev. Corr
LOCATION (Intercept) 2.3583 1.5357
YEAR96 0.6002 0.7747 -0.70
YEAR97 0.6735 0.8207 -0.80 0.46
Number of obs: 403, groups: LOCATION, 63
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.3273 0.2674 1.224 0.22083
YEAR96 1.3094 0.2542 5.152 2.58e-07 ***
YEAR97 -0.7578 0.2909 -2.605 0.00918 **
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Correlation of Fixed Effects:
(Intr) YEAR96
YEAR96 -0.746
YEAR97 -0.668 0.553
ただし、この推定は TICKS ~ YEAR + (1 | LOCATION) + (0 + YEAR | LOCATION)のように記述します。
> fit_glmm <- glmer(TICKS ~ YEAR + (1 | LOCATION) + (0 + YEAR | LOCATION), family="poisson", data=grouseticks)
> summary(fit_glmm)
Generalized linear mixed model fit by maximum likelihood (Laplace Approximation) ['glmerMod']
Family: poisson ( log )
Formula: TICKS ~ YEAR + (1 | LOCATION) + (0 + YEAR | LOCATION)
Data: grouseticks
AIC BIC logLik deviance df.resid
2268.7 2308.7 -1124.3 2248.7 393
Scaled residuals:
Min 1Q Median 3Q Max
-6.6442 -0.8112 -0.4797 0.8437 5.5779
Random effects:
Groups Name Variance Std.Dev. Corr
LOCATION (Intercept) 0.4151 0.6443
LOCATION.1 YEAR95 1.9431 1.3939
YEAR96 0.8755 0.9357 0.85
YEAR97 0.6006 0.7750 0.87 0.54
Number of obs: 403, groups: LOCATION, 63
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.3274 0.2674 1.224 0.22081
YEAR96 1.3094 0.2542 5.152 2.58e-07 ***
YEAR97 -0.7578 0.2909 -2.605 0.00918 **
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Correlation of Fixed Effects:
(Intr) YEAR96
YEAR96 -0.746
YEAR97 -0.668 0.553
また、TICKS ~ YEAR + (0 + YEAR | LOCATION)のように記述することでランダム効果を係数にのみ追加することも可能です。
> fit_glmm <- glmer(TICKS ~ YEAR + (0 + YEAR | LOCATION), family="poisson", data=grouseticks)
Warning message:
In checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
Model failed to converge with max|grad| = 0.0139508 (tol = 0.002, component 1)
> summary(fit_glmm)
Generalized linear mixed model fit by maximum likelihood (Laplace Approximation) ['glmerMod']
Family: poisson ( log )
Formula: TICKS ~ YEAR + (0 + YEAR | LOCATION)
Data: grouseticks
AIC BIC logLik deviance df.resid
2266.7 2302.7 -1124.3 2248.7 394
Scaled residuals:
Min 1Q Median 3Q Max
-6.6442 -0.8114 -0.4793 0.8432 5.5778
Random effects:
Groups Name Variance Std.Dev. Corr
LOCATION YEAR95 2.363 1.537
YEAR96 1.291 1.136 0.87
YEAR97 1.016 1.008 0.87 0.70
Number of obs: 403, groups: LOCATION, 63
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.3276 0.2676 1.224 0.22093
YEAR96 1.3099 0.2543 5.151 2.59e-07 ***
YEAR97 -0.7566 0.2908 -2.601 0.00929 **
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Correlation of Fixed Effects:
(Intr) YEAR96
YEAR96 -0.746
YEAR97 -0.669 0.554
optimizer (Nelder_Mead) convergence code: 0 (OK)
Model failed to converge with max|grad| = 0.0139508 (tol = 0.002, component 1)
R コード
ポアソン分布ん過分散を図示するRコードは以下になります。
> set.seed(1)
> hist(Y1 <- rpois(1000, 1), breaks=seq(0, 20), col="#FF00007F", freq=F, ylim=c(0, 0.8), las=1,
main="", xlab="X")
> hist(Y2 <- rpois(1000, 1 * exp(rnorm(1000, mean=0, sd=1))), col="#0000FF7F", add=T, freq=F, breaks=seq(0, 100))
> legend("right", legend=c("Poisson", "overdispersed Poisson"), pch=15, col=c("#FF00007F", "#0000FF7F"),
bty="n", cex=1.5)

参考