はじめに
この記事では、seabornライブラリからタイタニックのデータセットを使用して、ランダムフォレスト分類器を実装する方法を示します。
データセットの準備
最初に、必要なライブラリをインポートして、タイタニックのデータセットを読み込みます。
import numpy as np
import pandas as pd
import seaborn as sns
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import LabelEncoder
from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import accuracy_score, classification_report, confusion_matrix
import matplotlib.pyplot as plt
# Load the dataset
data = sns.load_dataset('titanic')
# Drop unnecessary columns
data = data.drop(['deck', 'embark_town', 'alive', 'who', 'adult_male', 'class'], axis=1)
# Handle missing values
data['age'] = data['age'].fillna(data['age'].median())
data['embarked'] = data['embarked'].fillna(data['embarked'].mode()[0])
# Encode categorical variables
encoder = LabelEncoder()
data['sex'] = encoder.fit_transform(data['sex'])
data['embarked'] = encoder.fit_transform(data['embarked'])
# Split the dataset into training and testing sets
X = data.drop('survived', axis=1)
y = data['survived']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=42)
モデルの構築
次に、scikit-learnを使用してランダムフォレスト分類器を作成します。
# 追加のハイパーパラメータを設定してランダムフォレスト分類器を作成
rf_clf = RandomForestClassifier(
n_estimators=100, # 決定木の数
criterion='gini', # 分割の品質を測定する関数('gini'または'entropy')
max_depth=None, # 決定木の最大の深さ(Noneは、すべての葉が純粋になるまでノードが展開されることを意味する)
min_samples_split=2, # 内部ノードを分割するために必要な最小限のサンプル数
min_samples_leaf=1, # 葉ノードに必要な最小限のサンプル数
max_features='auto', # 最適な分割を探す際に考慮する特徴量の数('auto'、'sqrt'、'log2'、または整数)
bootstrap=True, # ツリーを構築するときにブートストラップを使うかとうか
oob_score=False, # oobサンプルを使用して一般化精度を推定するかどうか
n_jobs=None, # fitおよびpredictに並列実行するジョブの数(-1はすべてのプロセッサを使用することを意味する)
random_state=42, # ブートストラップと特徴量サンプリングのランダム性を制御する
verbose=0, # フィットおよび予測時の冗長性を制御する
warm_start=False, # 前回のfit呼び出しの解を再利用し、アンサンブルにさらに推定子を追加する
class_weight=None # クラスの重み('balanced'またはNone)
)
以下に、追加のハイパーパラメータの簡単な説明を示します。
-
n_estimators
ランダムフォレストアンサンブル内の決定木の数を表します。つまり、個々の木が構築され、組み合わされる数を指定します。デフォルト値は100であり、ランダムフォレストは100個の決定木から構成されます。 -
criterion
分割の品質を測定するために使用される関数です。サポートされる基準は、ジニ不純度の場合は"gini"、情報利得の場合は"entropy"です。デフォルト値は"gini"に設定されています。 -
max_depth
決定木の最大深度を指定します。Noneの場合、ノードが全ての葉が純粋になるか、あるいは全ての葉がmin_samples_splitの数未満のサンプルを含むまで展開されます。値を高く設定すると過学習につながり、低く設定すると未学習につながる可能性があります。 -
min_samples_split
内部ノードを分割するために必要な最小限のサンプル数を指定します。この値を増やすと過学習を減らすことができますが、モデルの精度が低下する可能性があります。 -
min_samples_leaf
葉ノードに必要な最小限のサンプル数を指定します。この値を増やすと過学習を減らすことができますが、モデルの精度が低下する可能性があります。 -
max_features
最適な分割を探す場合に考慮する特徴量の数を指定します。'auto'、'sqrt'、'log2'、または整数に設定できます。'auto'の場合、max_features=sqrt(n_features)が使用されます。'sqrt'の場合、max_features=sqrt(n_features)が使用されます。'log2'の場合、max_features=log2(n_features)が使用されます。整数の場合、各分割で考慮される特徴量の数が考慮されます。 -
bootstrap
ツリーを構築するときにブートストラップサンプルを使用するかどうかを制御します。Falseの場合、全てのデータが使用されます。 -
oob_score
oobサンプルを使用して一般化精度を推定するかどうかを指定します。oobサンプルとは、特定のツリーのブートストラップサンプルに使用されなかったサンプルのことです。 -
n_jobs
fitおよびpredictに並列実行するジョブの数を指定します。-1は全てのプロセッサを使用することを意味します。 -
verbose
フィットおよび予測時の冗長性を制御します。値を高くすると、プロセス中により多くの情報が出力されます。 -
warm_start
Trueに設定すると、前回のfit呼び出しの解を再利用し、アンサンブルに新しい推定器を追加します。これは、ハイパーパラメータを反復的にチューニングするときに時間を節約するために役立ちます。前にトレーニングされた木を再利用し、新しい木を追加するため、全ての木を最初からトレーニングする必要がなくなります。 -
class_weight
クラスの重みを指定します。Noneの場合、全てのクラスが等しい重みを持つと想定されます。 'balanced'の場合、クラスの重みは各クラスのサンプル数に基づいて調整されます。これは、不均衡なデータセットを扱う場合に役立ちます。
トレーニングと評価
トレーニングデータに対してランダムフォレスト分類器をトレーニングし、テストデータでその性能を評価します。
# モデルのトレーニング
rf_clf.fit(X_train, y_train)
# テストセットで予測
y_pred = rf_clf.predict(X_test)
# モデルの評価
accuracy = accuracy_score(y_test, y_pred)
print(f"Accuracy: {accuracy:.2f}")
print("Classification Report:")
print(classification_report(y_test, y_pred))
print("Confusion Matrix:")
print(confusion_matrix(y_test, y_pred))
Accuracy: 0.78
Classification Report:
precision recall f1-score support
0 0.80 0.83 0.81 157
1 0.74 0.70 0.72 111
accuracy 0.78 268
macro avg 0.77 0.77 0.77 268
weighted avg 0.77 0.78 0.78 268
Confusion Matrix:
[[130 27]
[ 33 78]]
Feature Importanceの可視化
最後に、ランダムフォレストモデルのFeature Importanceを可視化します。
# Feature Importanceを計算する
importances = rf_clf.feature_importances_
# 重要性の高い順に特徴量をソートする
indices = np.argsort(importances)[::-1]
# 並び替えられた特徴量の名前を、ソートされたFeature Importanceに合わせて並べ替える
names = [X.columns[i] for i in indices]
# 棒グラフを作成する
plt.figure(figsize=(10, 5))
plt.title("Feature Importance")
plt.bar(range(X.shape[1]), importances[indices])
# 特徴量名をx軸のラベルとして追加
plt.xticks(range(X.shape[1]), names, rotation=90)
# プロットを表示する
plt.show()
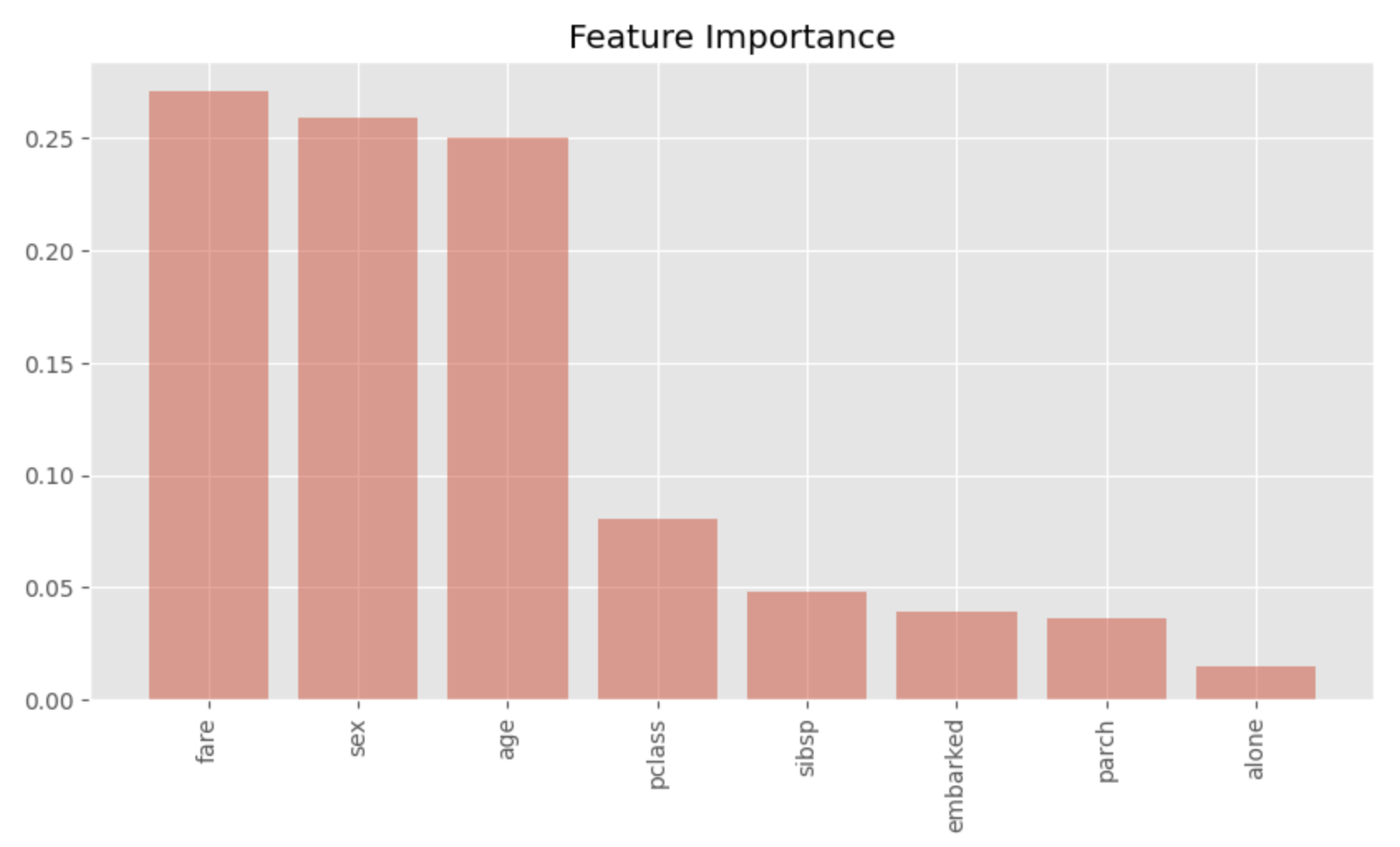
このようにプロットすることで、重要な特徴量が一目でわかります。sex、age、fareが非常に重要であることがわかります。これは説得力のある結果であり、タイタニック号での生死の違いをもたらした重要な要因です。