Apa itu Model Campuran linier Tergeneralisasi (GLMM)
Model Campuran linier Tergeneralisasi (Generalized Linear Mixed Model: GLMM) adalah model statistik yang merupakan pengembangan lebih lanjut dari GLM. Model ini dapat mewakili perbedaan individu dan regional yang tidak dapat diamati.
Perbedaan individu dan regional yang tidak dapat diamati termasuk, misalnya, berikut ini:
- Perbedaan individu dalam persepsi nyeri
Orang yang berbeda merespons secara berbeda terhadap nyeri yang sama. - Perbedaan individu dalam penilaian
Beberapa penilai lebih lunak daripada yang lain. - Perbedaan regional
Ada variasi regional dalam sifat nyeri.
Sebuah model yang tidak memperhitungkan perbedaan individu dan regional ini akan menyebabkan overdispersi. Overdispersi berarti bahwa distribusi probabilitas yang diasumsikan memiliki varians yang lebih besar daripada yang diasumsikan. Gambar berikut ini menunjukkan overdispersi dengan menggunakan distribusi Poisson sebagai contoh.
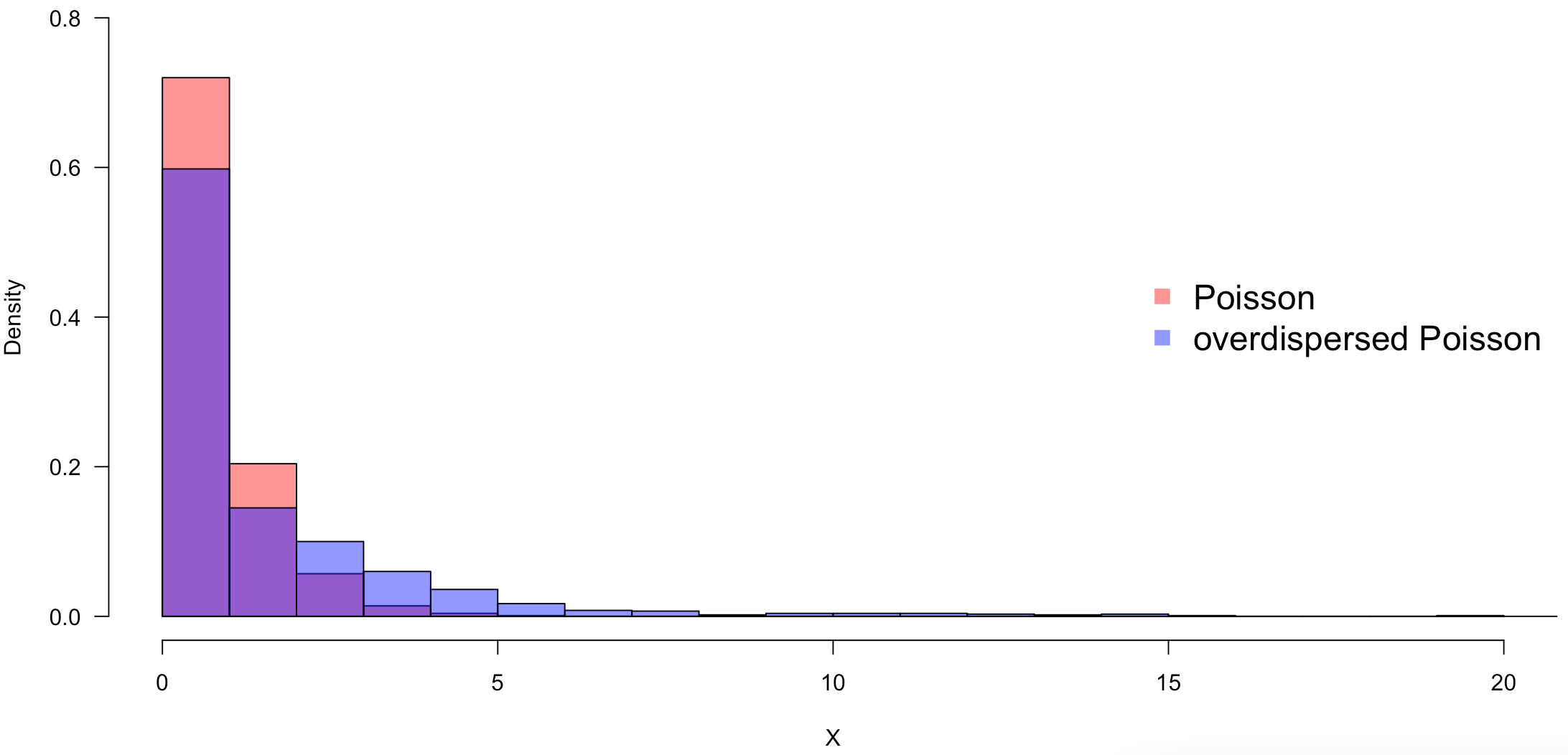
Perbedaan individu dan wilayah dapat menghasilkan distribusi Poisson yang lebih besar dari distribusi Poisson asli, seperti dalam kasus overdispersed Poisson, dan tanpa memperhitungkan perbedaan individu dan lokasi, data yang diamati tidak dapat dimodelkan dengan baik.
GLMM memperlakukan variasi yang tidak dapat dijelaskan seperti perbedaan individu dan lokasi sebagai efek acak dan memodelkannya seperti itu, dengan menentukan bahwa tidak mungkin untuk mengamati semua variabel penjelas.
Efek Acak
GLMM menambahkan efek acak ke prediktor linier. Misalnya, untuk individu
Kita asumsikan bahwa efek acak
Estimasi maksimum likelihood dari parameter akan dilakukan untuk
Kasus-kasus di mana GLMM diperlukan
Kriteria untuk menentukan apakah GLMM diperlukan adalah apakah Anda mengambil sampel individu atau lokasi yang sama berulang-ulang. Sebagai contoh, GLMM sering diterapkan pada data panel, yaitu data yang diamati berulang kali dari waktu ke waktu untuk individu.
Membangun GLMM
Mari kita membangun GLMM menggunakan dataset grouseticks. grouseticks adalah pengamatan jumlah kutu di kepala anak ayam belibis merah dan terdiri dari kolom-kolom berikut:
INDEX: (factor) chick number (observation level)TICKS: number of ticks sampledBROOD: (factor) brood numberHEIGHT: height above sea level (meters)YEAR: year (-1900)LOCATION: (factor) geographic location codecHEIGHT: centered height, derived from HEIGHT
Mari kita periksa data.
> require(lme4)
> library(lme4)
> data(grouseticks)
> summary(grouseticks)
INDEX TICKS BROOD HEIGHT YEAR LOCATION cHEIGHT
1 : 1 Min. : 0.00 606 : 10 Min. :403.0 95:117 14 : 24 Min. :-59.241
2 : 1 1st Qu.: 0.00 602 : 9 1st Qu.:430.0 96:155 4 : 20 1st Qu.:-32.241
3 : 1 Median : 2.00 537 : 7 Median :457.0 97:131 19 : 20 Median : -5.241
4 : 1 Mean : 6.37 601 : 7 Mean :462.2 28 : 19 Mean : 0.000
5 : 1 3rd Qu.: 6.00 643 : 7 3rd Qu.:494.0 50 : 17 3rd Qu.: 31.759
6 : 1 Max. :85.00 711 : 7 Max. :533.0 36 : 16 Max. : 70.759
(Other):397 (Other):356 (Other):287
> head(grouseticks)
INDEX TICKS BROOD HEIGHT YEAR LOCATION cHEIGHT
1 1 0 501 465 95 32 2.759305
2 2 0 501 465 95 32 2.759305
3 3 0 502 472 95 36 9.759305
4 4 0 503 475 95 37 12.759305
5 5 0 503 475 95 37 12.759305
6 6 3 503 475 95 37 12.759305
> plot(TICKS ~ HEIGHT, las=1)
Regresi Poisson tanpa efek acak
Kali ini kita akan membangun model regresi Poisson. Dengan menggunakan rata-rata jumlah kutu
dimana YEAR.
Kode berikut dapat digunakan untuk mengestimasi parameter.
> fit_glm <- glm(TICKS ~ YEAR, family=poisson(), data=grouseticks)
> summary)(fit_glm)
Call:
glm(formula = TICKS ~ YEAR, family = poisson(), data = grouseticks)
Deviance Residuals:
Min 1Q Median 3Q Max
-4.7110 -2.8888 -1.5183 0.7139 17.1467
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 1.78318 0.03790 47.05 <2e-16 ***
YEAR96 0.62348 0.04492 13.88 <2e-16 ***
YEAR97 -1.64109 0.08977 -18.28 <2e-16 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
(Dispersion parameter for poisson family taken to be 1)
Null deviance: 5847.5 on 402 degrees of freedom
Residual deviance: 4549.4 on 400 degrees of freedom
AIC: 5486.4
Number of Fisher Scoring iterations: 6
AIC adalah 5486.4.
Regresi Poisson dengan efek acak
mari kita buat model regresi Poisson dengan efek acak.
Model intersep acak
Tambahkan efek acak LOCATION dan nyatakan rata-rata jumlah kutu
Model ini juga disebut model intersep acak karena efek acak dimasukkan dalam intersep.
Anda dapat mengestimasi TICKS ~ YEAR + (1 | LOCATION).
> fit_glmm <- glmer(TICKS ~ YEAR + (1 | LOCATION), family="poisson", data=grouseticks)
> summary(fit_glmm)
Generalized linear mixed model fit by maximum likelihood (Laplace Approximation) ['glmerMod']
Family: poisson ( log )
Formula: TICKS ~ YEAR + (1 | LOCATION)
Data: grouseticks
AIC BIC logLik deviance df.resid
2306.3 2322.3 -1149.1 2298.3 399
Scaled residuals:
Min 1Q Median 3Q Max
-6.6399 -0.8844 -0.4708 0.8543 5.5757
Random effects:
Groups Name Variance Std.Dev.
LOCATION (Intercept) 1.644 1.282
Number of obs: 403, groups: LOCATION, 63
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.64992 0.18319 3.548 0.000388 ***
YEAR96 0.94371 0.08492 11.113 < 2e-16 ***
YEAR97 -1.43642 0.12298 -11.681 < 2e-16 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Correlation of Fixed Effects:
(Intr) YEAR96
YEAR96 -0.295
YEAR97 -0.241 0.520
AIC sekarang 2306.3, peningkatan yang signifikan dibandingkan model tanpa efek acak.
Varians dari efek acak untuk LOCATION adalah 1.644. Anda dapat memeriksa efek acak dari semua LOCATION dengan fungsi randf().
> ranef(fit_glmm)
$LOCATION
(Intercept)
1 1.24539318
2 0.92293852
3 -0.93190888
.
.
.
61 -1.35144088
62 -0.95446024
63 -1.15277383
with conditional variances for “LOCATION”
Nilai-nilai dari 1 sampai 63 adalah nilai LOCATION: intersep untuk 1 adalah 1.24539318 dan intersep untuk 3 adalah -0.93190888. Interpretasinya adalah bahwa LOCATION 1 relatif lebih banyak kutu daripada LOCATION 3.
Selain itu, efek acak INDEX dapat ditambahkan.
> fit_glmm <- glmer(TICKS ~ YEAR + (1 | LOCATION) + (1 | INDEX), family="poisson", data=grouseticks)
> summary(fit_glmm)
Generalized linear mixed model fit by maximum likelihood (Laplace Approximation) ['glmerMod']
Family: poisson ( log )
Formula: TICKS ~ YEAR + (1 | LOCATION) + (1 | INDEX)
Data: grouseticks
AIC BIC logLik deviance df.resid
1863.9 1883.9 -927.0 1853.9 398
Scaled residuals:
Min 1Q Median 3Q Max
-1.5003 -0.5933 -0.1154 0.2723 2.3743
Random effects:
Groups Name Variance Std.Dev.
INDEX (Intercept) 0.5081 0.7128
LOCATION (Intercept) 1.4679 1.2116
Number of obs: 403, groups: INDEX, 403; LOCATION, 63
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.2774 0.2107 1.316 0.188
YEAR96 1.2578 0.1821 6.906 4.97e-12 ***
YEAR97 -1.0856 0.2010 -5.400 6.67e-08 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Correlation of Fixed Effects:
(Intr) YEAR96
YEAR96 -0.536
YEAR97 -0.439 0.552
AIC sekarang 1863.9, yang merupakan peningkatan lebih lanjut.
Variansi efek acak untuk LOKASI adalah 1.4679 dan untuk INDEX adalah 0.5081. Kita dapat melihat bahwa efek acak dari INDEX lebih kecil daripada efek acak dari LOCATION.
Model koefisien acak
Mari kita tambahkan efek acak LOCATION tidak hanya pada intersep tetapi juga pada koefisien.
Model ini juga disebut model koefisien acak karena efek acak dimasukkan dalam koefisien.
Kode berikut dapat mengestimasi TICKS ~ YEAR + (YEAR | LOCATION).
> fit_glmm <- glmer(TICKS ~ YEAR + (YEAR | LOCATION), family="poisson", data=grouseticks)
> summary(fit_glmm)
Generalized linear mixed model fit by maximum likelihood (Laplace Approximation) ['glmerMod']
Family: poisson ( log )
Formula: TICKS ~ YEAR + (YEAR | LOCATION)
Data: grouseticks
AIC BIC logLik deviance df.resid
2266.7 2302.7 -1124.3 2248.7 394
Scaled residuals:
Min 1Q Median 3Q Max
-6.6442 -0.8112 -0.4797 0.8437 5.5779
Random effects:
Groups Name Variance Std.Dev. Corr
LOCATION (Intercept) 2.3583 1.5357
YEAR96 0.6002 0.7747 -0.70
YEAR97 0.6735 0.8207 -0.80 0.46
Number of obs: 403, groups: LOCATION, 63
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.3273 0.2674 1.224 0.22083
YEAR96 1.3094 0.2542 5.152 2.58e-07 ***
YEAR97 -0.7578 0.2909 -2.605 0.00918 **
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Correlation of Fixed Effects:
(Intr) YEAR96
YEAR96 -0.746
YEAR97 -0.668 0.553
Namun, estimasi ini adalah untuk model yang mengasumsikan korelasi antara TICKS ~ YEAR + (1 | LOCATION) + (0 + YEAR | LOCATION).
> fit_glmm <- glmer(TICKS ~ YEAR + (1 | LOCATION) + (0 + YEAR | LOCATION), family="poisson", data=grouseticks)
> summary(fit_glmm)
Generalized linear mixed model fit by maximum likelihood (Laplace Approximation) ['glmerMod']
Family: poisson ( log )
Formula: TICKS ~ YEAR + (1 | LOCATION) + (0 + YEAR | LOCATION)
Data: grouseticks
AIC BIC logLik deviance df.resid
2268.7 2308.7 -1124.3 2248.7 393
Scaled residuals:
Min 1Q Median 3Q Max
-6.6442 -0.8112 -0.4797 0.8437 5.5779
Random effects:
Groups Name Variance Std.Dev. Corr
LOCATION (Intercept) 0.4151 0.6443
LOCATION.1 YEAR95 1.9431 1.3939
YEAR96 0.8755 0.9357 0.85
YEAR97 0.6006 0.7750 0.87 0.54
Number of obs: 403, groups: LOCATION, 63
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.3274 0.2674 1.224 0.22081
YEAR96 1.3094 0.2542 5.152 2.58e-07 ***
YEAR97 -0.7578 0.2909 -2.605 0.00918 **
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Correlation of Fixed Effects:
(Intr) YEAR96
YEAR96 -0.746
YEAR97 -0.668 0.553
Hal ini juga memungkinkan untuk menambahkan efek acak hanya untuk koefisien dengan menulis TICKS ~ YEAR + (0 + YEAR | LOCATION).
> fit_glmm <- glmer(TICKS ~ YEAR + (0 + YEAR | LOCATION), family="poisson", data=grouseticks)
Warning message:
In checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
Model failed to converge with max|grad| = 0.0139508 (tol = 0.002, component 1)
> summary(fit_glmm)
Generalized linear mixed model fit by maximum likelihood (Laplace Approximation) ['glmerMod']
Family: poisson ( log )
Formula: TICKS ~ YEAR + (0 + YEAR | LOCATION)
Data: grouseticks
AIC BIC logLik deviance df.resid
2266.7 2302.7 -1124.3 2248.7 394
Scaled residuals:
Min 1Q Median 3Q Max
-6.6442 -0.8114 -0.4793 0.8432 5.5778
Random effects:
Groups Name Variance Std.Dev. Corr
LOCATION YEAR95 2.363 1.537
YEAR96 1.291 1.136 0.87
YEAR97 1.016 1.008 0.87 0.70
Number of obs: 403, groups: LOCATION, 63
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.3276 0.2676 1.224 0.22093
YEAR96 1.3099 0.2543 5.151 2.59e-07 ***
YEAR97 -0.7566 0.2908 -2.601 0.00929 **
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Correlation of Fixed Effects:
(Intr) YEAR96
YEAR96 -0.746
YEAR97 -0.669 0.554
optimizer (Nelder_Mead) convergence code: 0 (OK)
Model failed to converge with max|grad| = 0.0139508 (tol = 0.002, component 1)
Kode R
Kode R untuk menggambarkan overdispersi distribusi Poisson ditunjukkan di bawah ini.
> set.seed(1)
> hist(Y1 <- rpois(1000, 1), breaks=seq(0, 20), col="#FF00007F", freq=F, ylim=c(0, 0.8), las=1,
main="", xlab="X")
> hist(Y2 <- rpois(1000, 1 * exp(rnorm(1000, mean=0, sd=1))), col="#0000FF7F", add=T, freq=F, breaks=seq(0, 100))
> legend("right", legend=c("Poisson", "overdispersed Poisson"), pch=15, col=c("#FF00007F", "#0000FF7F"),
bty="n", cex=1.5)
